Vijenac 787
Kolumne
Mirko Planinić – Znanstveni zor
Umjetna inteligencija u potrazi za lijekovima
Zamislite da umjesto skupog eksperimenta možete računalom dizajnirati spojeve i proteine od kojih će nastati lijekovi. AlphaFold upravo je takav alat koji je nedavno iskoristio umjetnu inteligenciju za identifikaciju stotina tisuća potencijalnih psihodeličnih molekula – koje bi mogle pomoći u razvoju novih vrsta antidepresiva. Istraživanje po prvi put pokazuje da predviđanja AlphaFolda — dostupna pritiskom na gumb — mogu biti jednako korisna za otkrivanje lijekova kao i eksperimentalno izvedene proteinske strukture, za čije određivanje mogu biti potrebni mjeseci, pa čak i godine.
AlphaFold, alat umjetne inteligencije (UI), razvila je firma DeepMind iz Londona i time značajno unaprijedila strukturnu biologiju. Javna baza podataka AlphaFold sadrži predviđanja struktura za gotovo svaki poznati protein. Proteinske strukture molekula povezane s raznim bolestima koriste se u farmaceutskoj industriji za identifikaciju i poboljšanje obećavajućih lijekova. No mogu li predviđanja AlphaFolda zamijeniti zlatne standardne eksperimentalne modele u potrazi za novim lijekovima?
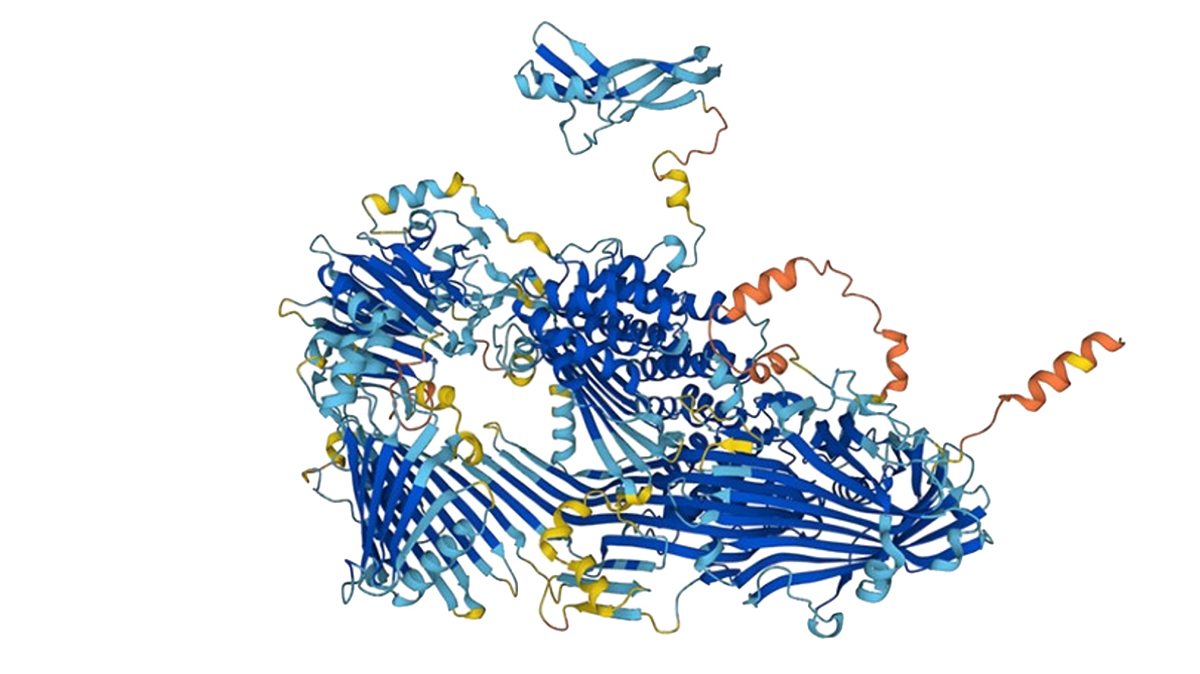
Strukture proteina koje je predvidio AlphaFold pomogle su u identificiranju spojeva kandidata za lijekove / Izvor tvrtka DeepMind
Više od deset studija otkrilo je da su predviđanja programa AplhaFold manje korisna od proteinskih struktura dobivenih rendgenskom kristalografijom kada se koristi za identifikaciju potencijalnih lijekova u metodi modeliranja koja se naziva spajanje protein-ligand.
Ovaj pristup — uobičajen u ranim fazama otkrivanja lijekova — uključuje modeliranje kako stotine milijuna kemikalija stupaju u interakciju s ključnim regijama ciljanog proteina, u nadi da će se identificirati spojevi koji mijenjaju aktivnost proteina. Prethodne studije pokazivale su da je umjetna inteligencija neefikasna u predviđanju struktura za upotrebu u lijekovima za koje je već poznato da se vežu na određeni protein. Znanstvenici, strukturni biolozi, sa Sveučilišta Sjeverne Karoline u Chapel Hillu, došli su do sličnog zaključka kada su usporedili AlphaFold strukture dvaju proteina povezanih s neuropsihijatrijskim stanjima u odnosu na poznate lijekove. Zapitali su se mogu li male razlike u odnosu na eksperimentalne strukture prouzročiti da predviđene strukture propuste određene spojeve koji se vežu na proteine, ali i omogućiti identifikaciju drugih koji nisu ništa manje obećavajući?
Kako bi se testirala ova ideja, tim istraživača koristio je eksperimentalne strukture dvaju proteina ne bi li virtualno pregledao stotine milijuna potencijalnih lijekova. Jedan protein, receptor koji osjeća neurotransmiter serotonin, prethodno je određen pomoću krioelektronske mikroskopije. Struktura drugog proteina, nazvanog σ-2 receptor, mapirana je kristalografijom X-zraka. Proveli su isto pretraživanje s modelima proteina izvađenih iz AlphaFold baze podataka. Zatim su sintetizirali stotine najperspektivnijih spojeva identificiranih s predviđenim i eksperimentalnim strukturama i izmjerili njihovu aktivnost u laboratoriju.
Istraživanja s predviđenim (AlphaFold) i eksperimentalnim strukturama dala su potpuno različite kandidate za lijekove. Dobivene molekule nisu ni sličile jedna drugoj. Ali na iznenađenje tima znanstvenika, udio označenih spojeva koji su stvarno promijenili aktivnost proteina na značajan način bio je gotovo identičan za dvije skupine. AlphaFold strukture identificirale su lijekove koji su najsnažnije aktivirali serotoninski receptor. Psihodelični lijek LSD djelomično djeluje na ovaj način, a mnogi istraživači traže nehalucinogene spojeve koji rade istu stvar, kao potencijalni antidepresivi. Ovo je uistinu novi rezultat i prekretnica u istraživanju. Znanstveno-istraživačka grupa Jensa Carlssona sa sveučilišta Uppsala iz Švedske otkrila je da su AlphaFold strukture dobre u identificiranju lijekova za traženu ciljanu klasu zvanu G-protein povezanih receptora, za koje je stopa uspješnosti bila oko 60%.
Polako raste optimizam i povjerenje u mogućnost predviđanja proteinskih struktura za otkrivanje lijekova. Eksperimentalno određivanje struktura nije trivijalno, a mnogi potencijalni spojevi možda neće biti dostupni postojećim eksperimentalnim alatima. Bilo bi vrlo zgodno kad bismo mogli pritisnuti gumb i dobiti strukturu koju možemo koristiti za otkrivanje liganda. Ligandi sadrže jedan ili više atoma nemetala koji posjeduju slobodni elektronski par (O, S, N, P, C, Cl) i doniraju ga u kovalentnu vezu sa središnjim atomom (najčešće metalnim kationom).
Predviđene strukture korisne su za neke vrste lijekova, ali ne i za druge, a nije uvijek jasno što je primjenjivo. U oko 10% slučajeva predviđanja koja AlphaFold smatra vrlo točnima bitno se razlikuju od eksperimentalne strukture. Čak i kada predviđene strukture mogu pomoći u identificiranju tragova prema lijekovima, često su potrebni detaljniji eksperimentalni modeli kako bi se optimizirala svojstva određenog kandidata za lijek.
Neke procjene govore da bi u otprilike jednoj trećini slučajeva AlphaFold struktura mogla pokrenuti projekt za novi lijek što je ušteda od nekoliko godina u odnosu na sadašnju proceduru.
Cilj DeepMindove spin-off tvrtke Isomorphic Labs za otkrivanje lijekova u Londonu upravo je to preskakanje nekoliko godina eksperimenata koje bi radili da nema AlphaFolda.
Tvrtka kaže da će u istraživanju koristiti novu verziju AlphaFolda koja može predvidjeti strukture proteina kada su vezani za lijekove i druge molekule u interakciji.
Takvi alati neće u potpunosti zamijeniti eksperimente, ali njihov potencijal za pomaganje u pronalaženju novih lijekova ne treba zanemariti. Strukturni biolozi i dalje će biti potrebni u timovima za istraživanje lijekova, ali umjetna inteligencija mijenja njihov način rada koji postaje kreativniji.
787 - 9. svibnja 2024. | Arhiva
Klikni za povratak






